
18 July 2022
El cerdo Ibérico es una raza reconocida a nivel mundial por sus productos curados de elevada calidad organoléptica. Su alimentación exclusiva con bellotas y pastos en la dehesa durante la montanera en la última etapa de su vida, desde los 100 a los 160 kilogramos de peso, está estrechamente relacionada con esta calidad.
Estado de la mejora genética
Los sistemas de cría tradicionales utilizados en el cerdo Ibérico, hasta hace muy pocos años alejados de los criterios productivistas normales en otras razas, y su orientación comercial hacia un sector de la población muy concreto, mantuvo lo mantuvo alejado de los programas de mejora aplicados comúnmente desde hace bastantes décadas en muchas de la razas porcinas europeas y americanas más extendidas.
Es más, hasta hace pocos años era frecuente encontrar opiniones acerca de lo innecesario de la mejora genética de la raza.
El enorme crecimiento de los últimos 30 años de la población de cerdos Ibéricos y la generalización del consumo de sus productos, con una modificación sustancial de los sistemas de cría inclinándolos mayoritariamente hacia el cruce con Duroc y la intensificación de las producciones, trajo también consigo el interés de determinados ganaderos e industriales por la mejora genética de determinados aspectos productivos que fueran acompasando las mejoras en manejo y alimentación que poco a poco se iban implementado.
Por ello, los programas de mejora en cerdos Ibéricos son un fenómeno reciente, están poco extendidos, y se han llevado a cabo, hasta ahora, al margen de las grandes compañías de genética mundiales.
Y además, su aplicación genérica es compleja, puesto que la variedad de sistemas de producción (montanera vs intensivo o puros vs cruzados) y de intereses comerciales dificultan la implementación de un programa único para el conjunto de la población.
Figura 1. La demanda de productos del cerdo ibérico ha promovido el desarrollo de los programas de mejora

La mayor parte de la producción de cerdos Ibéricos es cruzada con Duroc, por lo que la mejora de los caracteres de crecimiento y composición de la canal se consigue mediante el cruce con líneas de esta raza ya seleccionadas por las multinacionales de genética.
Por otro lado, para la mejora de los caracteres maternos como el tamaño de camada, dado el bajo valor de su heredabilidad, se requiere el control reproductivo de un número muy elevado de cerdas, lejos del tamaño de población habitual en las explotaciones de Ibérico, y una estructura piramidal muy jerárquica.
Este requisito deja en manos de muy pocas ganaderías la posibilidad de abordar esta mejora con cierta probabilidad de respuesta que justifique el esfuerzo realizado. Son ganaderías mayoritariamente con manejos intensivos, orientados a la producción cruzada y que, en este caso, sí suelen implementar sus programas con la ayuda de empresas especializadas.
Los programas de mejora exclusivos para cerdos Ibéricos puros cebados en sistema extensivos (cebo de campo o montanera) son minoritarios
Los objetivos de selección tradicionales en las otras razas porcinas comerciales, como la velocidad de crecimiento o el porcentaje de magro, no pueden ser tratados en ellos de la misma forma puesto que, por un lado, la norma de calidad que regula los productos establece una edad mínima al sacrificio y, por otro, los largos procesos de curación que soportan los jamones y paletas requieren tanto de una suficiente de cobertura de grasa subcutánea como de una infiltración de grasa intramuscular apreciable.
Por ello, en los programas de mejora para el cerdo Ibérico, casi prácticamente desde sus inicios, se consideró la conveniencia de incluir caracteres de calidad de carne como el porcentaje de grasa intramuscular, o de grasa como el perfil de ácidos grasos, puesto que los programas debían de tener en cuenta la especificidad de estos productos (Silió, 2000).
Por otro lado, la rusticidad o el mantenimiento de la adaptación del animal al medio de dehesa y a sus condiciones climáticas, también debería ser considerada en un futuro de manera definitiva.
Desde el punto de vista técnico, la mayoría de los programas de mejora convencionales se basan en el denominado modelo animal, que integra la matriz con todas las relaciones de parentesco conocidas, y en evaluaciones genéticas BLUP (Best linear unbiased prediction o mejor predicción lineal insesgada).
Con esta metodología se puede utilizar la información de campo de distintos sexos, granjas, condiciones ambientales, etc.; obteniéndose estimas de los valores mejorantes (EBV, estimated breeding values) corregidos o ajustados para estos factores externos.
Los animales con mejores EBV pasan a integrarse en el elenco de reproductores de la piara o rebaño, por lo que en varias generaciones se obtiene una respuesta a la selección o progreso genético.
Sin embargo, existen diversas limitaciones o ineficiencias de esta metodología que pueden ser corregidas y resueltas por las técnicas de genética molecular.
El enorme desarrollo de la biología molecular en las últimas décadas del siglo XX ha influido también de manera definitiva en el campo de la mejora genética animal.
La información molecular permitiría seleccionar aquellos individuos que sean los mejores para el carácter objeto de selección, sin necesidad de cuantificarlo, de medirlo
La base de su aplicación recae en los que denominamos polimorfismo, es decir, una variación en la secuencia del ADN que puede ser un cambio puntual (SNP, Single Nucleotide Polymorphism) en una de las letras (bases nitrogenadas: A, C, G y T) que componen el ADN, una pérdida o ganancia de un fragmento de la secuencia (deleción/inserción) o un mayor o menor número de repeticiones de dos o tres de las citadas bases (microsatélite).
Teniendo en cuenta esto, si para un carácter de interés tenemos entre diferentes individuos una variación (genotipo) que está asociada a un determinado fenotipo (medida de ese carácter), podremos seleccionar aquellos individuos que tengan el genotipo para el mejor valor del fenotipo.
Figura 2. Los caracteres de calidad de carne se seleccionan utilizando el enfoque molecular

Los caracteres de calidad de la carne son propicios para ser abordados desde este enfoque molecular, puesto que en general son costosos o difíciles de medir y, de obtener éxito, un incremento en el valor de estos productos gracias a su mejora en calidad justificaría el esfuerzo económico que estas técnicas requieren
A continuación se resumen algunos de los antecedentes y se refieren experiencias más recientes en la aplicación información molecular a programas de mejora para calidad de carne en cerdo Ibérico, fundamentalmente orientados a la cría y engorde en pureza en sistemas extensivos tradicionales.
Antecedentes moleculares
El antecedente más importante de los estudios de marcadores moleculares asociados a caracteres de calidad de carne en cerdo Ibérico parte del consorcio IBMAP en el cual investigadores de diferentes universidades y centros de investigación (Universidad Autónoma de Barcelona, IRTA de Cataluña e INIA, entre otros), participaron en siete proyectos de investigación desarrollados entre los años 1996 y 2018, en los que se llevaron a cabo distintos estudios que analizaban la base genética de caracteres de calidad de carne como la grasa intramuscular y el espesor de grasa dorsal (Óvilo et al., 2000), la composición de ácidos grasos (Clop et al., 2003) y el pH y color de la carne (Óvilo et al., 2002) en un cruce experimental Ibérico x Landrace.
En estos trabajos se detectaron regiones del genoma asociadas a los caracteres citados (denominados QTL, de quantitative trait loci, o locus asociado a un carácter cuantitativo) y se propusieron genes candidatos que pudiesen contener polimorfismos potenciales de ser aplicados en programas de mejora de calidad de carne, como por ejemplo el Receptor de la leptina (LEPR), la Acil-coA sintentasa (ACSL4), la Acetil-coA carboxilasa (ACACA) o la transferasa microsomal de triglicéridos (MTTP) (Óvilo et al., 2005; Mercadé et al., 2006; Muñoz et al., 2007; Estellé et al., 2009).
A partir de los resultados de estos trabajos y de otras experiencias en otras razas porcinas, se llevaron a cabo otros estudios específicos en el cerdo Ibérico.
Fernández et al. (2008) observaron que un polimorfismo localizado en el gen que codifica para la proteína citocromo c oxidasa estaba asociado con el contenido en grasa intramuscular y el contenido en proteína.
Asimismo, Muñoz et al. (2011) indicaron que un polimorfismo localizado en el gen LEPR estaba asociado con grasa intramuscular, perfil de ácidos grasos, espesor de grasa dorsal y el color de la carne en cerdos cruzados Duroc x Ibérico, de tal forma que proponían este polimorfismo para seleccionar verracos Duroc.
Otro ejemplo es el del gen que codifica para la desaturasa de ácidos grasos (SCD), en el cual la combinación de distintos polimorfismos puede ser usada para seleccionar individuos con un mejor potencial de desaturación y contenido en grasa intramuscular, factores determinantes de la calidad de carne y que también pueden ser implementados en el cruce Duroc x Ibérico (Fernandez et al., 2017).
Todos estos estudios fueron muy útiles y valiosos, pero desde el punto de vista práctico han sido pocos los polimorfismos detectados en genes candidatos que hayan podido ser finalmente implementados, de una manera práctica, en programas de selección en Ibérico.
Figura 3. Los polimorfismos detectados en los genes candidatos no se ha utilizado en la mejora genética práctica en ibérico

Técnicas de secuenciación masiva
A mediados de la primera década del siglo XXI se desarrollan las llamadas técnicas de ‘secuenciación masiva’ que permiten secuenciar el genoma de los individuos a gran velocidad y a un coste muy bajo teniendo en cuenta la gran cantidad de información obtenida. Utilizando estas técnicas, los trabajos realizados en los últimos años se han centrado principalmente en tres de ellas:
- Genotipado masivo de SNP repartidos por todo el genoma y análisis de asociación entre estos genotipos y los fenotipos de los individuos. Este tipo de análisis tiene una potencia mayor que los estudios anteriores, ya que estos utilizaban un número sustancialmente menor de marcadores. Por lo tanto, las regiones cromosómicas concernidas están más delimitadas, permitiendo una detección más precisa del polimorfismo asociado al carácter.
- Análisis del transcriptoma utilizando la técnica conocida como RNAseq. El transcriptoma es el conjunto de genes que se están expresando dentro de un tejido en un momento puntual del individuo. Es decir, son todas las moléculas de ARN producidas por el ADN que se corresponden con un determinado tejido, una determinada raza, o un estado fisiológico, dieta, edad, etc. Por lo tanto, el transcriptoma va a ser variable en función de todos estos factores. Los estudios de RNAseq se basan principalmente en el análisis de la expresión diferencial entre grupos distintos o extremos. Pueden existir muchos tipos de análisis en función de los grupos de comparación, como por ejemplo comparaciones entre fenotipos extremos, entre estirpes o razas distintas o análisis del efecto de la nutrición u otro factor externo (tratamientos sanitarios, temperatura incluso manejo o instalaciones).
- Finalmente, el uso de toda esta información de secuenciación masiva ha permitido llevar a cabo otro tipo de estudios, más aplicados, en los que se preselecciona un número más reducido de SNP que afectan a genes más concretos que se haya visto en estos trabajos previos que tiene relación o asociación con los caracteres de interés. En este caso, se analiza si SNP que afectan a los caracteres de calidad de carne pueden tener una implementación práctica, ya sobre un terreno más ganadero, en los programas de mejora.
Análisis de asociación genómico
El análisis de asociación a nivel genómico (conocido como GWAS, Genome-wide association analysis study) consiste en el análisis estadístico de asociación entre cientos de miles de SNP a lo largo del genoma y los caracteres de interés. La representación típica de los resultados es con un gráfico denominado Manhattan Plot (Figura 4) en el cual aparece una serie de SNP demarcando regiones genómicas que están asociados estadísticamente con el carácter analizado.
Figura 4. Manhattan Plot con la región de asociación significativa con grasa intramuscular (resultados propios, aún sin publicar)
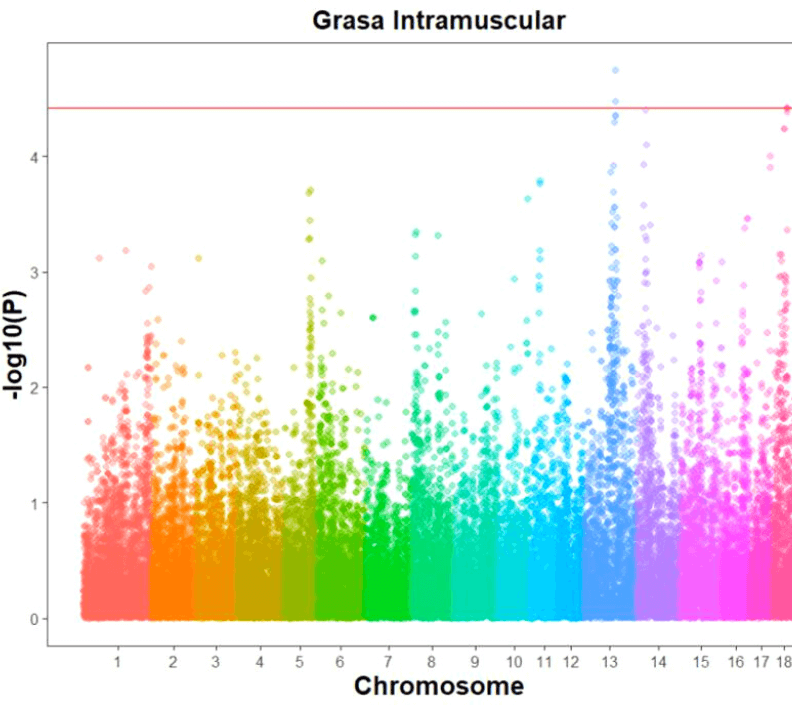
Hasta la fecha no hay muchos estudios de asociación genómica en caracteres de calidad de carne en Ibérico puro o en los cruces experimentales con otras razas de cerdo magro
Dentro del consorcio IBMAP se hicieron una serie de investigaciones de asociación genómica en los que se detectaron regiones asociadas a la composición de ácidos grasos y al contenido de grasa intramuscular en cruces Ibérico x Duroc, Ibérico x Landrace e Ibérico x Pietrain (Ramayo-Caldas et al., 2012; Muñoz et al., 2013; Martínez-Montes et al., 2018 y Crespo-Piazuelo et al., 2020).
En cerdos ibéricos puros, hasta el momento sólo hay una publicación científica de un análisis de la composición de la grasa de distintas estirpes de cerdo Ibérico (Pena et al., 2019), si bien existen algunas iniciativas en marcha que se espera puedan generar resultados sobre una amplia gama de caracteres de calidad en próximos meses.
Los estudios de asociación genómica se suelen hacer a partir de la información del genotipado con paneles de SNPs comerciales. Hasta la fecha hay diferentes paneles disponibles: SNP60 v2 Genotyping BeadChip de Illumina con 64.232 SNPs (Figura 2), GGP-Porcine HD de Neogen con 70.231 y Axiom Porcine Genotyping Array de Affymetrix con 658.692 SNPs.
En el caso de los dos primeros, el principal inconveniente que tienen es que no se tuvo en cuenta la raza Ibérica para su diseño, por lo que hay mucha información que no se puede utilizar. En el tercer caso sí se tuvo en cuenta la raza Ibérica, pero al tener una cantidad muy superior de SNPs su coste es más elevado, por lo que es complicado poder financiar este tipo de estudios en los que se requiere, además, genotipar una cantidad grande de individuos.
Figura 5. Genotyping BeadChip de Illumina con 64.232 SNPs

Análisis de expresión diferencial del transcriptoma
Son varios los trabajos realizados mediante la secuenciación del transcriptoma (RNAseq). Los primeros de ellos también correspondían con los cruces experimentales del consorcio IBMAP (Ramayo-Caldas et al., 2012b; Corominas et al., 2013; Puig-Oliveras et al., 2014), principalmente enfocados al análisis de expresión diferencial en distintos tejidos de cerdos con valores extremos para composición de ácidos grasos, tanto de grasas subcutánea e intramuscular, y también para contenido en grasa intramuscular.
Posteriormente, se han llevado a cabo estudios en cerdos Ibéricos puros de montanera (Muñoz et al., 2018b; Fernández Barroso et al., 2020b) analizando la expresión del transcriptoma en lomo para individuos extremos para caracteres como grasa intramuscular, terneza y contenido en mioglobina (en proceso de publicación) que está relacionado a su vez con el color de la carne.
El objetivo de estos últimos ha sido fundamentalmente detectar genes concretos que se estén expresando de manera diferencial en los grupos de individuos con fenotipos extremos, para poder posteriormente buscar polimorfismos en estos genes e incorporar esta información al programa de mejora.
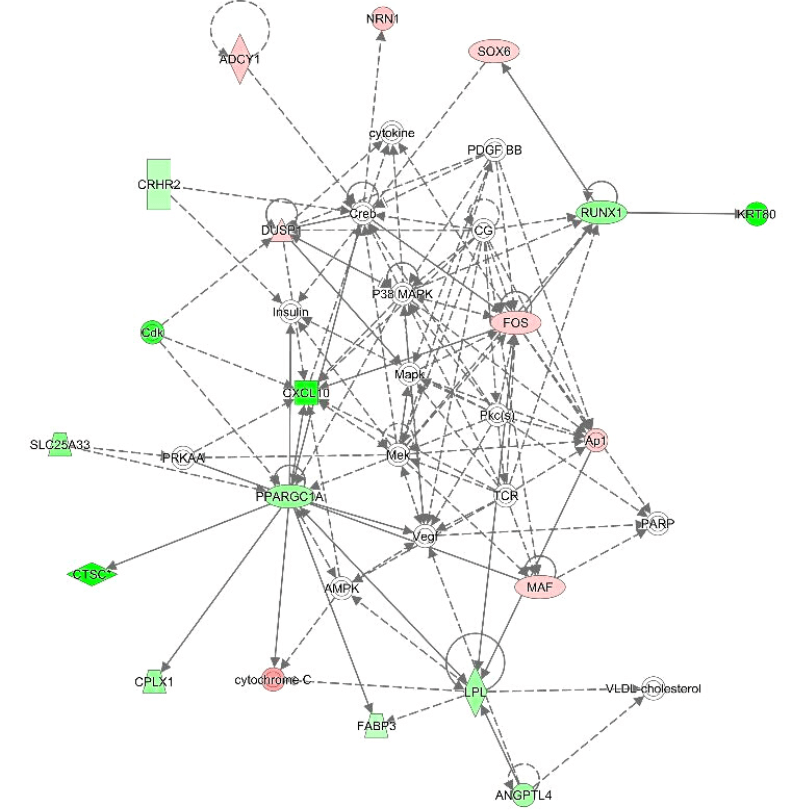
Además, los resultados de estos trabajos permiten profundizar en el conocimiento de las rutas biológicas implicadas en la definición del carácter estudiado (Figura 3).
Con respecto a los estudios de transcriptoma, hay que indicar finalmente que se han publicado en cerdos Ibéricos varios trabajos que no están directamente relacionados con programas de mejora, sino con comparaciones entre Ibérico y su cruce con Duroc (Óvilo et al., 2014; Ayuso et al., 2015), con expresión diferencial para tratamientos con dietas experimentales (Benítez et al., 2015) o comparaciones entre distintas estirpes (Villaplana-Velasco et al., 2021).
Figura 6. Interacción de genes diferencialmente expresados entre grupos de animales con carnes más tiernas y más duras (Fernández-Barroso et al., 2020b)
Paneles de SNPs
A partir de la información obtenida en los estudios de asociación genómica y de expresión diferencial se han llevado a cabo diseños de paneles específicos de SNPs para un carácter en particular o para un grupo de caracteres.
En el caso de resultados de GWAS, se pueden utilizar los mismos SNPs que se han identificado o buscar SNPs en genes próximos a éstos. Estos genes se seleccionan según su funcionalidad, es decir, si dan lugar a proteínas que tengan una función relacionada con el carácter en cuestión.
Por otro lado, también a partir de los estudios de expresión diferencial del transcriptoma se pueden seleccionar genes candidatos que estén diferencialmente expresados y que estén funcionalmente relacionados con el carácter. Además es factible la búsqueda de polimorfismos dentro de estos genes y comprobar si los grupos de expresión diferencial opuesta tienen genotipos distintos para estos polimorfismos.
En la actualidad existen dos programas de mejora en los que participa el Centro de I+D en cerdo Ibérico de Zafra, que están experimentando con paneles de SNPs, uno de 32 y otro de 64, diseñados por el grupo de porcino de INIA para varios caracteres como grasa intramuscular, terneza, capacidad de retención de agua o color.
Los resultados de éstos son bastante prometedores y han mostrado como polimorfismos localizados en los genes para la enzima calpaina 1 (CAPN1) y para una subunidad de la proteína quinasa (PRKAG3), pueden ser utilizados para seleccionar verracos y obtener una descendencia mejorada para caracteres relacionados con la terneza de la carne (Fernández-Barroso et al., 2020a).
Se ha visto que un polimorfismo localizado en el gen que codifica para la hormona adiponectina (ADIPOQ) podría utilizarse para mejorar el contenido en grasa intramuscular
Por último, utilizando los resultados de los trabajos previos (Muñoz et al 2018a, 2018b; Fernández-Barroso et al., 2020b) se ha diseñado un nuevo panel de 32 SNPs específico para una ganadería comercial orientada a la producción de cerdos Ibéricos de montanera.
Este panel puede ser usado tanto como diagnóstico para descartar la entrada de posibles reproductores de fuera de esta población que puedan tener una composición genética no deseada, como para contribuir a la selección en un futuro de los candidatos de la propia piara.
A día de hoy, se están finalizando los análisis de comprobación para poder implementar una utilización rutinaria del panel es esta población.
Otras aplicaciones de ciencias -ómicas en cerdo Ibérico. Bienestar y sostenibilidad.
En este siglo XXI nos enfrentamos a una serie de desafíos medioambientales que van a requerir una adaptación de las especies ganaderas en dos sentidos.
Por un lado, prepararlos para que ellos mismos sean capaces de dar respuesta a estos desafíos (resiliencia). Por otro, tratar de conseguir mejoras fisiológicas que minimicen la huella de la producción animal sobre el medioambiente (sostenibilidad).
El cerdo Ibérico, a pesar de su adaptación al entorno y a su menor efecto sobre el medio, al menos durante el cebo en montanera, no se debería quedar al margen de estos retos
Uno de los problemas ambientales más obvios es el efecto que tiene el aumento de la temperatura que se viene produciendo en los últimos años, tanto en la media como en la máxima anual, sobre los individuos.
El efecto del estrés térmico, conocido y descrito ya en producción animal (Hoffman et al., 2010), es su incidencia sobre la fisiología de los animales, alterando tanto aspectos productivos como reproductivos.
Los cerdos Ibéricos se han adaptado durante siglos a las altas temperaturas y a la escasez de recursos durante las épocas estivales en el suroeste de la península Ibérica. Sin embargo, en los últimos años los ganaderos están observando algunas anomalías reproductivas que pueden ser atribuidas a una duración cada vez mayor del verano, con repetidos registros máximos de temperatura.
Estos problemas, tratados como caracteres que pueden ser objeto de selección, son muy apropiados para ser abordado con las llamadas metodologías o ciencias -ómicas.
Así por ejemplo, la información molecular u -ómica puede ser utilizada para analizar cómo el estrés térmico afecta a los cerdos Ibéricos detectando biomarcadores, sustancias que nos permiten monitorizar un determinado estado biológico. En la actualidad el Departamento de Mejora Genética Animal del INIA desarrolla un proyecto cuyo objetivo es encontrar estos biomarcadores para estrés térmico en reproductores machos y hembras, utilizando técnicas de metabolómica, transcriptómica y metagenómica.
Ya hemos visto anteriormente qué es el análisis del transcriptoma. De manera análoga, el análisis del metaboloma permite determinar los metabolitos, sustancias concretas producidas o consumidas por el metabolismo, que hay en un tejido o fluido (sangre, por ejemplo) determinado del individuo en un momento concreto. Finalmente, el análisis del metagenoma permite determinar la composición de una comunidad microbiana, también en un tejido o fluido en concreto. Si podemos asociar cualquiera de estos elementos (gen, metabolito o categoría microbiana) a animales con mayor resiliencia a estrés térmico, podremos seleccionar aquellos individuos que estén mejor preparados para su tolerancia en un futuro.
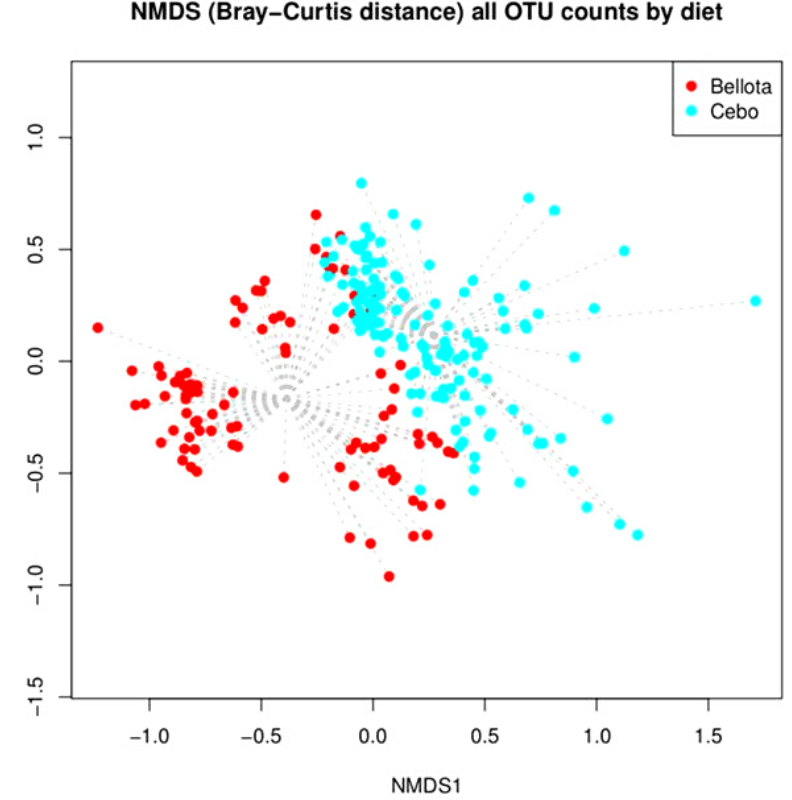
La última de estas ciencias -ómicas, la metagenómica, está siendo muy desarrollada en diferentes especies ganaderas, ya que se ha comprobado como cambios en la composición de las comunidades microbianas en diferentes localizaciones (fundamentalmente en el tracto digestivo, aunque también en el excretor, reproductor, piel, pulmones) están relacionados con cambios en caracteres productivos y reproductivos. No sólo eso, con los estudios metagenómicos se está investigando sobre la potencial aplicación de productos pre- y probióticos que reduzcan el uso de antibióticos en un futuro, así como su uso como herramienta predictora del tipo de alimentación de los individuos. Como otro ejemplo de la utilización de estas técnicas en cerdo Ibérico, se ha visto que caracterizando la comunidad microbioma del intestino se puede detectar fraudes en la alimentación del mismo durante el cebo (García-Casco et al., 2018, Figura 4).
Figura 7. Agrupación de cerdos Ibéricos según su alimentación. Se pueden distinguir dos grupos en función de la alimentación de bellota o montanera y cebo. (García-Casco et al., 2018)
En definitiva, a pesar de la incorporación tardía del cerdo Ibérico a los programas de mejora genética convencionales, en los últimos años la aplicación de las nuevas técnicas de la genética molecular a variados aspectos productivos en esta raza se ha incrementado considerablemente, con varios grupos de investigación nacionales interesados en ella.
Los hallazgos encontrados y los progresos efectuados en caracteres de calidad de carne o su enorme utilidad para afrontar los retos ambientales y climáticos actuales, justifican por sí mismos los esfuerzos realizados y la inversión futura en este campo del conocimiento.
Bibliografía
- Ayuso M. et al. 2015. Comparative analysis of muscle transcriptome between pig genotypes identifies genes and regulatory mechanisms associated to growth, fatness and metabolism. PLoS One 10 (12), e0145162.
- Benítez R. et al. 2015. Effects of dietary fat saturation on fatty acid composition and gene transcription in different tissues of Iberian pigs. Meat Sci. 102, 59-68.
- Clop A et al., 2003. Detection of QTL affecting fatty acid composition in the pig. Mamm. Genome 14, 650-656.
- Corominas J et al., 2013. Analysis of porcine adipose tissue transcriptome reveals differences in de novo fatty acid synthesis in pigs with divergent muscle fatty acid composition. BMC genomics 14(1), 1-14.
- Crespo-Piazuelo D et al., 2020. Identification of strong candidate genes for backfat and intramuscular fatty acid composition in three crosses based on the Iberian pig. Sci. rep. 10(1), 1-17.
- Estellé J et al., 2009. A non-synonymous mutation in a conserved site of the MTTP gene is strongly associated with protein activity and fatty acid profile in pigs. Anim. Genet. 40(6), 813-820.
- Fernández A.I. et al., 2008. Mitochondrial genome polymorphisms associated with longissimus muscle composition in Iberian pigs. J. Anim. Sci. 86(6), 1283-1290.
- Fernández A.I. et al., 2017. Validating porcine SCD haplotype effects on fatty acid desaturation and fat deposition in different genetic backgrounds. Livest. Sci. 205, 98-105.
- Fernández-Barroso M.A. et al. 2020a. Genetic parameter estimation and gene association analyses for meat quality traits in open‐air free‐range Iberian pigs. J. Anim. Breed. Genet. 137:581–598.
- Fernández-Barroso M.A. et al. 2020b. Differences in the loin tenderness of Iberian pigs explained through dissimilarities in their transcriptome expression profile. Animals 10, 1715.
- García-Casco J.M. et al. 2018. Major differences in gut microbiota composition of Iberian pigs in montanera vs commercial systems. 69th EAAP Ann. Meet., Dubrovnik.
- Hoffmann I. 2010. Climate change and the characterization, breeding and conservation of animal genetic resources. Anim. Genet. 41 (1), 32–46.
- Martinez-Montes A.M. et al., 2018. Using genome wide association studies to identify common QTL regions in three different genetic backgrounds based on Iberian pig breed. PLoS One, 13(3), e0190184.
- Mercadé A. et al., 2006. Characterization of the porcine acyl-CoA synthetase long-chain 4 gene and its association with growth and meat quality traits. Anim. Genet. 37, 219-224.
- Muñoz G. et al., 2007. QTL detection on porcine chromosome 12 for fatty‐acid composition and association analyses of the fatty acid synthase, gastric inhibitory polypeptide and acetyl‐coenzyme a carboxylase alpha genes. Anim. Genet. 38(6), 639-646.
- Muñoz G. et al., 2011. Effects of porcine MC4R and LEPR polymorphisms, gender and Duroc sire line on economic traits in Duroc x Iberian crossbred pigs. Meat Sci. 88: 169-173
- Muñoz M. et al., 2013. Genome-wide analysis of porcine backfat and intramuscular fat fatty acid composition using high-density genotyping and expression data. BMC genomics 14(1), 1-15.
- Muñoz M. et al., 2018a. Diversity across major and candidate genes in European local pig breeds. PLoS One 13(11), e0207475.
- Muñoz M. et al., 2018b. Identification of candidate genes and regulatory factors underlying intramuscular fat content through longissimus dorsi transcriptome analyses in heavy Iberian pigs. Frontiers in genetics 9, 608.
- Ovilo C. et al. 2000. A QTL for intramuscular fat and backfat thickness is located on porcine chromosome 6. Mamm. Genome 11, 344-346.
- Ovilo C. et al. 2002. Quantitative trait locus mapping for meat quality traits in an Iberian x Landrace F2 pig population. J. Anim. Sci. 80, 2801-2808.
- Ovilo C. et al. 2005. Fine mapping of porcine chromosome 6 QTL and LEPR effects on body composition in multiple generations of an Iberian by Landrace intercross. Genet. Res. 85, 57-67.
- Óvilo C. et al. 2014. Longissimus dorsi transcriptome analysis of purebred and crossbred Iberian pigs differing in muscle characteristics. BMC Genomics 15,413.
- Pena R.N. et al. 2019. Five genomic regions have a major impact on fat composition in Iberian pigs. Sci. Rep. 9, 2031.
- Puig-Oliveras A. et al., 2014. Differences in muscle transcriptome among pigs phenotypically extreme for fatty acid composition. PloS One 9(6), e99720.
- Ramayo-Caldas Y. et al., 2012a. Genome-wide association study for intramuscular fatty acid composition in an Iberian× Landrace cross. J. Anim. Sci. 90(9), 2883-2893.
- Ramayo-Caldas Y. et al., 2012b. Liver transcriptome profile in pigs with extreme phenotypes of intramuscular fatty acid composition. BMC genomics 13(1), 1-18.
- Silió L., 2000. Iberian pig breeding programme. ICAR Technical Series 3. Workshop Bella, Italy.
- Vilaplana-Velasco A. et al. 2021. Comparative transcriptome profile between Iberian pig varieties provides new insights into their distinct fat deposition and fatty acids content. Animals 11 (3), 627.






