
01 November 2024
Uno de los mayores desafíos de la agricultura es enfrentar condiciones cambiantes como plagas, enfermedades, sequías y otros factores que pueden reducir el rendimiento de los cultivos.
Para abordar estos problemas se han desarrollado tecnologías capaces de mejorar las capacidades de las plantas con el objetivo de hacerlas más resilientes y capacitarlas para enfrentarse a los efectos del cambio climático.
Durante la generación de nuevas variedades, la selección de estas es uno de los puntos más críticos del proceso, tanto por los recursos como por el tiempo que consume. Una de las herramientas más utilizadas en este paso son los marcadores moleculares, lo que se conoce como selección asistida por marcadores.
Esta técnica es sencilla una vez se ha identificado el marcador y hace posible el análisis de numerosas líneas a bajo coste. Pese a los avances en secuenciación masiva, que permiten conocer todo el genoma (secuencia completa de ADN) de una planta, tanta información no suele ser práctica en programas de mejora, por lo que para centrarse en características individuales se suele trabajar con marcadores.
Un marcador molecular es una molécula que da información sobre el individuo, puede ser de diferente tipología, pero los más importantes en el ámbito de la mejora vegetal son los marcadores moleculares genéticos.
Estos marcadores son variaciones en una misma secuencia de ADN (Figura 1) de la planta que pueden ser usadas para identificar rasgos de interés agrícola, como la resistencia a enfermedades, tolerancia a la sequía o calidad del fruto.
Además, pueden llegar a asociarse con una probabilidad de heredarla. También puede entenderse el marcador como una herramienta, basada en las variantes que presenta dicha secuencia.

En un proceso tradicional de mejora, las plantas son seleccionadas tras observar su crecimiento y respuesta a la exposición de distintos factores (ej. estrés biótico o abiótico), según las necesidades del mejorador. Si bien la anterior metodología es igualmente válida, con los marcadores moleculares es posible identificar las características genéticas desde una fase muy temprana, ahorrando tiempo y recursos, puesto que permite trabajar desde inicio con plantas con un mayor potencial genético.
En términos más específicos, y sin tener en cuenta la influencia ambiental y los rasgos complejos, los marcadores pueden ayudar a inferir el fenotipo de la planta, que son aquellas cualidades observables, a través del genotipo (el ADN) de ella.
Cómo desarrollar y utilizar un marcador molecular
Imaginemos que queremos desarrollar un marcador de arroz asociado a la resistencia de salinidad. A grandes rasgos, los principales pasos serían los siguientes:
1- Obtener germoplasma para estudiar el rasgo deseado. Si queremos mejorar la resistencia al estrés salino y no hay descritas variedades con estas características, el primer paso es encontrar plantas de arroz que ya sean potencialmente más tolerantes. Es mucho más probable que las puedas localizar en zonas con suelos salinos donde se cultiven variedades tradicionales, ya que estas se habrán adaptado poco a poco a esas condiciones. También es posible obtener candidatos de especies silvestres o, en el mejor de los casos, de líneas que ya tengan esta resistencia descrita, directamente.
2- Análisis del ADN. Se compara entonces la secuencia del ADN de líneas candidatas con otras que no lo son (Figura 2). Todas las diferencias que aparezcan son potenciales marcadores. Si lo que nos interesa es encontrar uno específicamente asociado a la resistencia de la salinidad, tendremos que valorar numerosos genotipos para acotar lo máximo posible la relación entre la resistencia a la salinidad y la variabilidad genética. En este paso puede ser de gran utilidad estudiar marcadores para la resistencia a la salinidad que ya estén descritos en otras plantas, sin necesidad de que estén estrechamente emparentadas con el arroz. Aquí también cobra importancia el concepto del mapeo genético que, entre otros aspectos, ayuda a estimar la probabilidad de que una característica de la planta esté ligada a un cambio en el ADN y su heredabilidad.
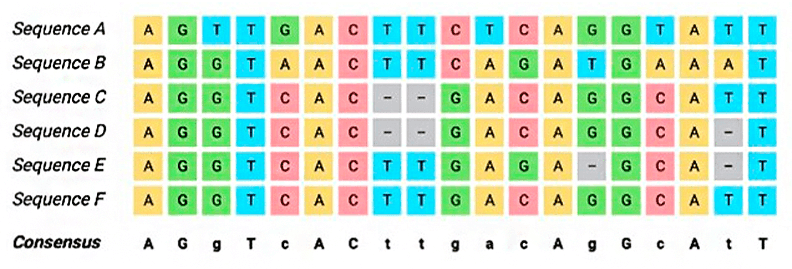
3- Selección de candidatos. Se valorará la secuencia de ADN de todas las líneas que tengamos en nuestra colección y se seleccionarán como candidatos aquellas que tengan el marcador de interés, o con cambios en zonas del ADN cercanas al mismo. Hay que tener presente que el marcador podrá ser utilizado en cualquier planta de la misma especie y, en muchos casos, en plantas del mismo género o familia.
4- Ensayos de validación. Aquellas plantas candidatas se evaluarán en diferentes condiciones de estrés salino y se observará su comportamiento. Esto permitirá validar la asociación entre el marcador y el rasgo deseado.
5-Desarrollo del marcador. Una vez identificado el cambio del ADN asociado a esa resistencia, se crea el marcador. Esto es, se le da un nombre y se describe ampliamente, desde la secuencia, localización, utilidad, etc. Es posible patentar el uso de un marcador, y muchas empresas y centros de investigación lo hacen. No obstante, al menos a escala europea, es importante destacar que una secuencia de ADN en sí misma no se puede patentar, pero si un uso concreto. Es decir, lo que se podrá patentar en este caso es el proceso de cómo utilizar el marcador para seleccionar plantas de arroz resistentes a salinidad. Aquí tenéis un ejemplo de una patente para para la resistencia al virus del rugoso en tomate.
Cualquier nuevo programa de mejora vinculado a la resistencia a la salinidad en arroz e incluso en otros cereales podrá partir directamente desde el paso 3.
Tipos de marcadores moleculares genéticos
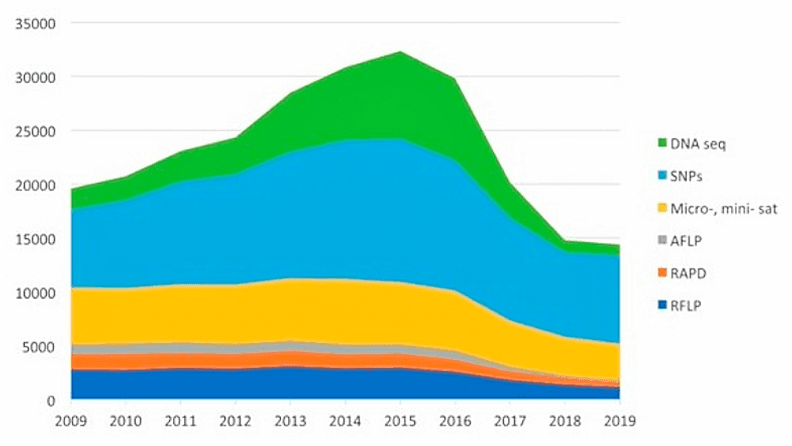
Existen muchos tipos de marcadores genéticos (Figura 3) y se pueden clasificar en función de la naturaleza del cambio en el ADN o la forma en la que se identifican (Figura 3). Si bien todos son relevantes, los que actualmente están cobrando más importancia en mejora vegetal son los SNPs (Polimorfismos de Nucleótido Simple, comúnmente llamados Esnips).
Hay que tener presente que, independiente del marcador, estos no siempre se comportan como un 'todo o nada', sino que pueden favorecer o estar asociados a una característica.
Por ejemplo, en caso de plagas y enfermedades, en lugar de asociarse a una resistencia completa a la planta, puede que esta tenga una mayor probabilidad de ser tolerante.

Una aclaración
Si conocéis de cerca el entorno de mejora vegetal es probable que hayáis escuchado también términos como alelo y QTL en el mismo contexto que marcadores. Los términos 'marcador genético/molecular' suelen entremezclarse con 'QTL' y 'alelos', pero no son lo mismo.
El marcador es una herramienta, el QTL es una zona del ADN y el alelo es una “versión” del gen. Es decir, un alelo es una de las variantes de un gen.
Un locus de rasgo cuantitativo (QTL) es una región de ADN que afecta un rasgo cuantitativo (ej. peso, longitud). Estos rasgos, además, suelen estar controlados por distintas zonas del genoma, no solo una secuencia en particular.
Un marcador genético o molecular es una variación de una secuencia y no tiene por qué influir directamente en un rasgo.
Si dentro de un gen puede variar la secuencia de ADN, ese gen tendrá alelos diferentes para cada cambio. Las secuencias que pueden variar serán marcadores. Si además ese gen influye en una característica cuantitativa, esa zona del genoma será un QTL.
En conclusión
Si se pretende disminuir el uso de insumos químicos y que exista una seguridad alimentaria global, es imprescindible que las plantas ya presenten “de fábrica” la mayor capacidad de respuesta a los desafíos sociales y ambientales posible. Es decir, necesitamos cultivos resilientes.
En este sentido, los marcadores moleculares permiten desarrollar cultivos de forma más eficiente, contribuyendo desde la selección temprana de genotipos hasta la creación de nuevas variedades para enfrentar los desafíos actuales y futuros de la agricultura.





